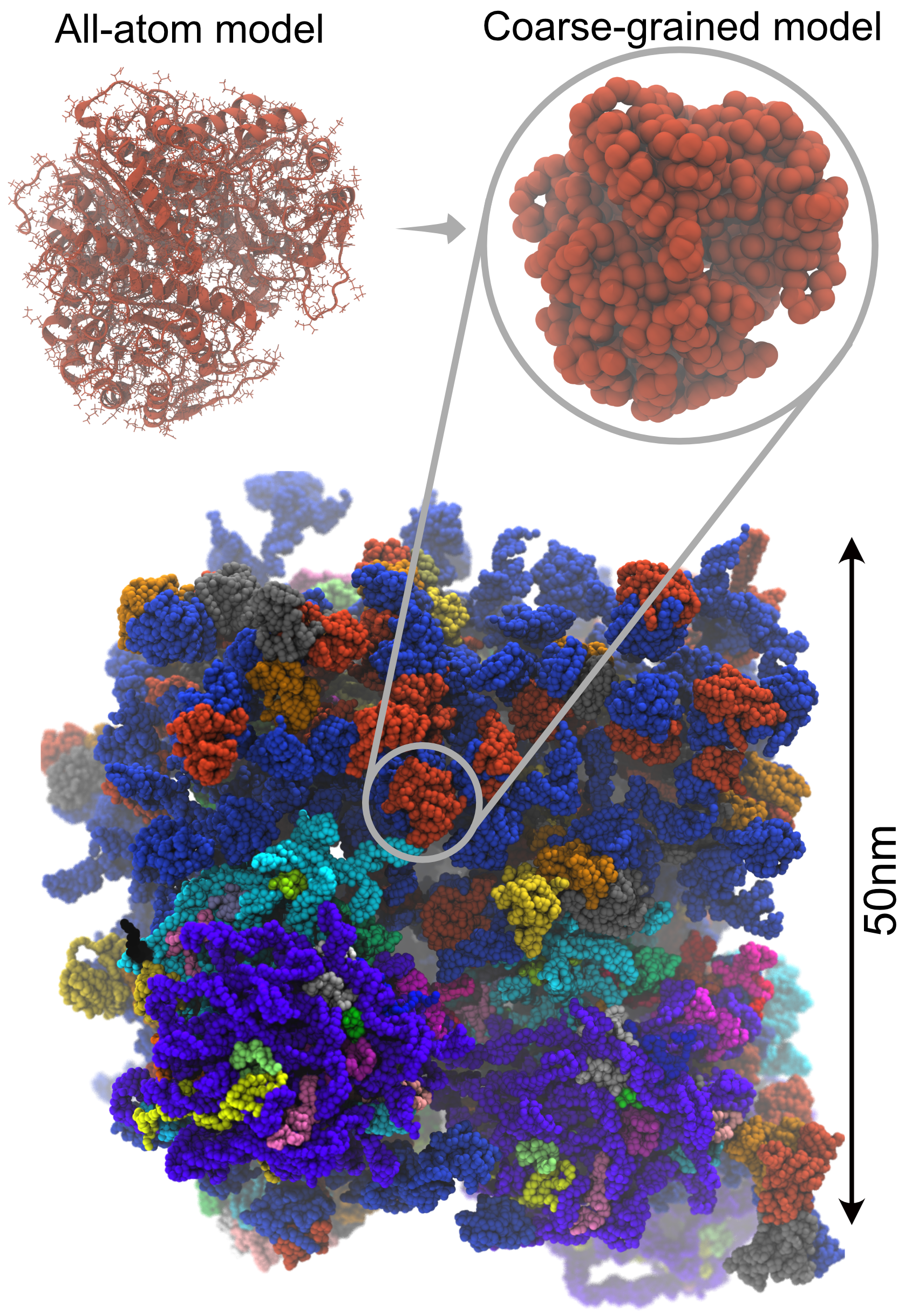
細胞内では、タンパク質、核酸、脂質などの様々な高分子が含まれており、それぞれに相互作用しています。このような環境で、生命活動に必要な機能がどのように働くのかはとても重要な問題です。様々な種類の分子が互いを認識することで、反応性や安定性を変化させていると考えられています。しかし、様々な高分子を含む環境の研究は、膨大な計算量が必要となり、困難でした。私たちは、シミュレーションが観測可能な時間・空間的な範囲を広げるべく、様々な粗視化モデルの開発を行っています。全原子モデル、量子化学手法であるQM/MMも含め、解像度の異なる分子モデルを有機的につないだマルチレゾリューション手法の開発を進め、GENESISに実装しています。スーパーコンピュータ「富岳」を用いて、様々な分子が含まれる細胞スケールシミュレーションを目指します。
参考文献
- “Scaling Molecular Dynamics Beyond 100,000 Processor Cores for Large-Scale Biophysical Simulations”, Jaewoon Jung, Wataru Nishima, Marcus Daniels, Gavin Bascom, Chigusa Kobayashi, Adetokunbo Adedoyin, Michael Wall, Anna Lappala, Dominic Phillips, William Fischer, Chang-Shung Tung, Tamar Schlick, Yuji Sugita, and Karissa Y. Sanbonmatsu, J. Comput. Chem., 40, 1919-1930 (2019).
- “Biomolecular interactions modulate macromolecular structure and dynamics in atomistic model of a bacterial cytoplasm”, Isseki Yu, Takaharu Mori, Tadashi Ando, Ryuhei Harada, Jaewoon Jung, Yuji Sugita, and Michael Feig., eLife, 5, e19274 (2016).
- “Domain Motion Enhanced (DoME) Model for Efficient Conformational Sampling of Multidomain Proteins”, Chigusa Kobayashi, Yasuhiro Matsunaga, Ryotaro Koike, Motonori Ota, and Yuji Sugita, J. Phys. Chem. B, 119, 14584-14593 (2015).
共同研究者
Michael Feig (Michigan State University)、Karissa Sanbonmatsu (Los Alamos National Laboratory), Shoji Takada (京都大学)